Análisis de brotes en tiempo real: el ébola como estudio de caso - parte 1
Introducción
Esta práctica (en tres partes) simula la evaluación temprana y la reconstrucción de un brote de enfermedad por el virus del Ébola (EVE). Introduce varios aspectos del análisis de la etapa inicial de un brote, incluida la tasa de letalidad (CFR por sus siglas en inglés de Case Fatality Ratio), curvas epidemiológicas (parte 1), estimación de la tasa de crecimiento, datos del rastreo de contactos, retrasos y estimaciones de transmisibilidad (parte 2), así como la reconstrucción de la cadena de transmisión mediante el uso de outbreaker2 (parte 3).
Nota: Esta práctica se deriva de las prácticas Ebola simulation part 1: early outbreak assessment y Ebola simulation part 2: outbreak reconstruction
Resultados del aprendizaje
Al final de esta práctica, usted será capaz de:
Cargar y limpiar datos de brotes en R (parte 1)
Estimar la tasa de letalidad (CFR) (parte 1)
Calcular y graficar la incidencia de la base de datos de casos (parte 1)
Estimar e interpretar la tasa de crecimiento y el tiempo de duplicación de la epidemia (parte 2)
Estimar el intervalo de serie a partir de los datos pareados de infectadas / personas infectadas (parte 2)
Estimar e interpretar el número de reproducción de la epidemia (parte 2)
Pronóstico de la incidencia futura a corto plazo (parte 2)
Reconstruir quién infectó a quién utilizando datos epidemiológicos y genéticos (parte 3)
Un nuevo brote de EVE en un país ficticio de África occidental
Se ha notificado un nuevo brote de EVE en un país ficticio de África occidental. El Ministerio de Salud se encarga de coordinar la respuesta al brote, y lo ha contratado como consultor en análisis epidémico para informar la respuesta en tiempo real.
Paquetes necesarios
Los siguientes paquetes, disponibles en CRAN o github, son necesarios para este análisis. Para instalarlos, ejecute los siguientes códigos:
# install.packages("remotes")
# install.packages("readxl")
# install.packages("outbreaks")
# install.packages("incidence")
# remotes::install_github("reconhub/epicontacts@ttree")
# install.packages("distcrete")
# install.packages("epitrix")
# remotes::install_github("annecori/EpiEstim")
# remotes::install_github("reconhub/projections")
# install.packages("ggplot2")
# install.packages("magrittr")
# install.packages("binom")
# install.packages("ape")
# install.packages("outbreaker2")
# install.packages("here")Una vez instalados los paquetes, es posible que deba abrir una nueva sesión de R. Ahora cargue las bibliotecas de la siguiente manera:
library(readxl)
library(outbreaks)
library(incidence)
library(epicontacts)
library(distcrete)
library(epitrix)
library(EpiEstim)
library(projections)
library(ggplot2)
library(magrittr)
library(binom)
library(ape)
library(outbreaker2)
library(here)
library(tidyverse)Datos iniciales (lectura de datos en R)
Se le ha proporcionado la siguiente base de datos de casos (linelist en inglés) y datos de contacto:
linelist_20140701.xlsx: una base de datos de casos que contiene información de casos hasta el 1 de julio de 2014; y
contact_20140701.xlsx: una lista de contactos reportados por los casos hasta el 1 de julio de 2014. “infector” indica una fuente potencial de infección y “case_id” con quién se tuvo el contacto.
Para leer en R, descargue estos archivos y use la función read_xlsx()
del paquete readxl para importar los datos. Cada grupo de datos
importados creará una tabla de datos almacenada como el objeto tibble.
- Llame primero la
linelist, y - después los
contacts.
Por ejemplo, su primera línea de comando podría verse así:
linelist <- read_excel("data/linelist_20140701.xlsx", na = c("", "NA"))Tómese su tiempo para mirar los datos y la estructura aquí.
- ¿Son los datos y el formato similares a bases de datos de casos que ha visto en el pasado?
- Si fuera parte del equipo de investigación de un brote, ¿qué otra información le gustaría recopilar?
## [1] 169 11
## # A tibble: 6 x 11
## case_id generation date_of_infection date_of_onset date_of_hospitalisation
## <chr> <dbl> <chr> <chr> <chr>
## 1 d1fafd 0 NA 2014-04-07 2014-04-17
## 2 53371b 1 2014-04-09 2014-04-15 2014-04-20
## 3 f5c3d8 1 2014-04-18 2014-04-21 2014-04-25
## 4 6c286a 2 NA 2014-04-27 2014-04-27
## 5 0f58c4 2 2014-04-22 2014-04-26 2014-04-29
## 6 49731d 0 2014-03-19 2014-04-25 2014-05-02
## # ... with 6 more variables: date_of_outcome <chr>, outcome <chr>,
## # gender <chr>, hospital <chr>, lon <dbl>, lat <dbl>
Tenga en cuenta que para análisis posteriores, deberá asegurarse de que
todas las fechas estén almacenadas correctamente como Date objects.
Puede hacer esto usando la función as.Date, por ejemplo:
linelist$date_of_onset <- as.Date(linelist$date_of_onset, format = "%Y-%m-%d")Los datos ahora deberían verse así:
## # A tibble: 6 x 11
## case_id generation date_of_infection date_of_onset date_of_hospitalisation
## <chr> <dbl> <date> <date> <date>
## 1 d1fafd 0 NA 2014-04-07 2014-04-17
## 2 53371b 1 2014-04-09 2014-04-15 2014-04-20
## 3 f5c3d8 1 2014-04-18 2014-04-21 2014-04-25
## 4 6c286a 2 NA 2014-04-27 2014-04-27
## 5 0f58c4 2 2014-04-22 2014-04-26 2014-04-29
## 6 49731d 0 2014-03-19 2014-04-25 2014-05-02
## # ... with 6 more variables: date_of_outcome <date>, outcome <chr>,
## # gender <chr>, hospital <chr>, lon <dbl>, lat <dbl>
## # A tibble: 6 x 3
## infector case_id source
## <chr> <chr> <chr>
## 1 d1fafd 53371b other
## 2 f5c3d8 0f58c4 other
## 3 0f58c4 881bd4 other
## 4 f5c3d8 d58402 other
## 5 20b688 d8a13d funeral
## 6 2ae019 a3c8b8 other
Limpieza de datos y análisis descriptivo
Mire más de cerca los datos contenidos en este linelist.
- ¿Qué observa?
## # A tibble: 6 x 11
## case_id generation date_of_infection date_of_onset date_of_hospitalisation
## <chr> <dbl> <date> <date> <date>
## 1 d1fafd 0 NA 2014-04-07 2014-04-17
## 2 53371b 1 2014-04-09 2014-04-15 2014-04-20
## 3 f5c3d8 1 2014-04-18 2014-04-21 2014-04-25
## 4 6c286a 2 NA 2014-04-27 2014-04-27
## 5 0f58c4 2 2014-04-22 2014-04-26 2014-04-29
## 6 49731d 0 2014-03-19 2014-04-25 2014-05-02
## # ... with 6 more variables: date_of_outcome <date>, outcome <chr>,
## # gender <chr>, hospital <chr>, lon <dbl>, lat <dbl>
## [1] "case_id" "generation"
## [3] "date_of_infection" "date_of_onset"
## [5] "date_of_hospitalisation" "date_of_outcome"
## [7] "outcome" "gender"
## [9] "hospital" "lon"
## [11] "lat"
Puede notar que faltan entradas. Un paso importante en el análisis es identificar cualquier error en la entrada de datos. Aunque puede ser difícil evaluar los errores en los nombres de los hospitales, es de esperar que la fecha de la infección sea siempre anterior a la fecha de aparición de los síntomas.
Limpie este conjunto de datos para eliminar cualquier entrada con períodos de incubación negativo o de 0 días.
## identificar errores en la entrada de datos (período de incubación negativo)
mistakes <-
mistakes
linelist[mistakes, ]## [1] 46 63 110
## # A tibble: 3 x 11
## case_id generation date_of_infection date_of_onset date_of_hospitalis~
## <chr> <dbl> <date> <date> <date>
## 1 3f1aaf 4 2014-05-18 2014-05-18 2014-05-25
## 2 ce9c02 5 2014-05-27 2014-05-27 2014-05-29
## 3 7.000000000000~ 6 2014-06-10 2014-06-10 2014-06-16
## # ... with 6 more variables: date_of_outcome <date>, outcome <chr>,
## # gender <chr>, hospital <chr>, lon <dbl>, lat <dbl>
Guarde su base de datos de casos “limpia” como un objeto nuevo:
linelist_clean
linelist_clean <- linelist[-mistakes, ]¿Qué otras fechas negativas o errores podría querer verificar si tuviera el conjunto de datos completo?
Calculemos la tasa de letalidad (CFR)
Aquí está el número de casos por estado de resultado. ¿Cómo calcularía el CFR a partir de esto?
table(linelist_clean$outcome, useNA = "ifany")##
## Death Recover <NA>
## 60 43 63
Piense en qué hacer con los casos cuyo resultado es NA.
n_dead <- sum(linelist_clean$outcome %in% "Death")
n_known_outcome <- sum(linelist_clean$outcome %in% c("Death", "Recover"))
n_all <- nrow(linelist_clean)
cfr <- n_dead / n_known_outcome
cfr_wrong <- n_dead / n_all
cfr_with_CI <- binom.confint(n_dead, n_known_outcome, method = "exact")
cfr_wrong_with_CI <- binom.confint(n_dead, n_all, method = "exact")Miremos las curvas de incidencia
La primera pregunta que queremos saber es simplemente: ¿qué tan mal está? El primer paso del análisis es descriptivo: queremos dibujar una epicurva o curva epidemiológica. Esto permite visualizar la incidencia a lo largo del tiempo por fecha de inicio de los síntomas.
Usando el paqueteincidence calcular la incidencia diaria a partir del
linelist_clean basado en las fechas de inicio de los síntomas.
Almacene el resultado en un objeto llamado i_daily; el resultado
debería verse así:
i_daily## <incidence object>
## [166 cases from days 2014-04-07 to 2014-06-29]
##
## $counts: matrix with 84 rows and 1 columns
## $n: 166 cases in total
## $dates: 84 dates marking the left-side of bins
## $interval: 1 day
## $timespan: 84 days
## $cumulative: FALSE
plot(i_daily, border = "black")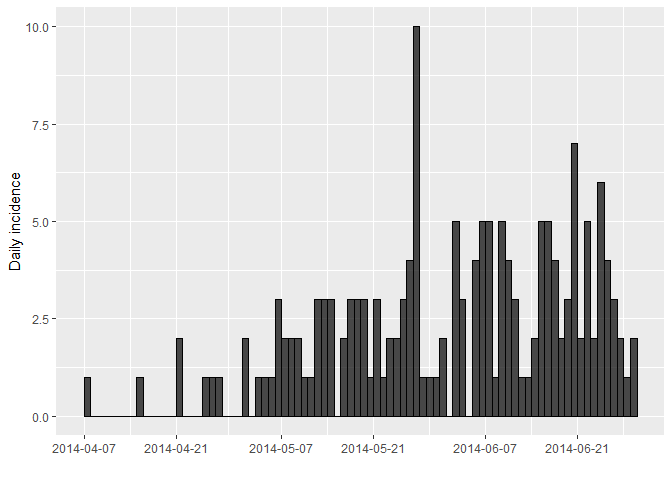
Es posible que observe que las fechas de incidencia i_daily$dates se
detienen en la última fecha en la que tenemos datos sobre la fecha de
inicio de los síntomas (29 de junio de 2014). Sin embargo, una
inspección minuciosa de la base de datos de casos muestra que la última
fecha (de cualquier entrada) es, de hecho, un poco posterior (1 de julio
de 2014). Puede usar el argumento last_date en la función incidence
para cambiar esto.
## <incidence object>
## [166 cases from days 2014-04-07 to 2014-07-01]
##
## $counts: matrix with 86 rows and 1 columns
## $n: 166 cases in total
## $dates: 86 dates marking the left-side of bins
## $interval: 1 day
## $timespan: 86 days
## $cumulative: FALSE
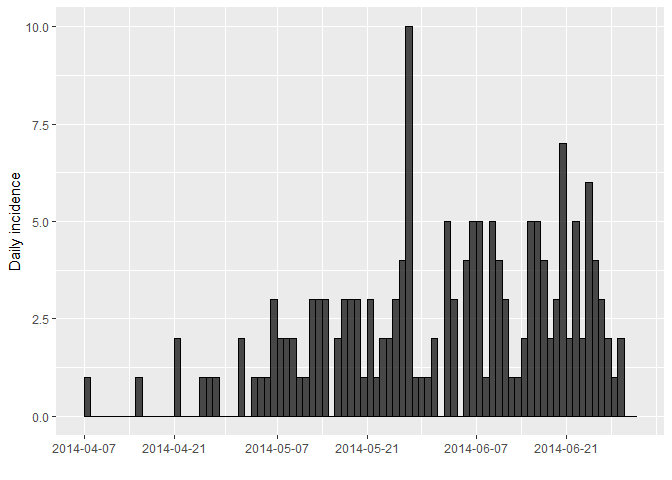
Otro problema es que puede ser difícil interpretar las tendencias al
observar la incidencia diaria, por lo que también calcule y grafique la
incidencia semanal i_weekly, como se ve a continuación:
i_weekly <- incidence(linelist_clean$date_of_onset, interval = 7,
last_date = as.Date(max(linelist_clean$date_of_hospitalisation, na.rm = TRUE)))
i_weekly## <incidence object>
## [166 cases from days 2014-04-07 to 2014-06-30]
## [166 cases from ISO weeks 2014-W15 to 2014-W27]
##
## $counts: matrix with 13 rows and 1 columns
## $n: 166 cases in total
## $dates: 13 dates marking the left-side of bins
## $interval: 7 days
## $timespan: 85 days
## $cumulative: FALSE
plot(i_weekly, border = "black")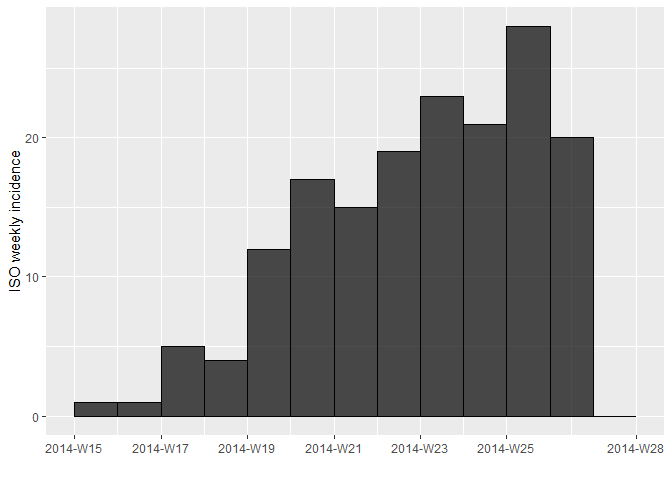
Guardar datos y resultados
Este es el final de la práctica de la parte 1. Antes de pasar a la parte 2, deberá guardar los siguientes objetos:
dir.create("data/clean") # cree un directorio de datos limpio si no existe
saveRDS(i_daily, "data/clean/i_daily.rds")
saveRDS(i_weekly, "data/clean/i_weekly.rds")
saveRDS(linelist, "data/clean/linelist.rds")
saveRDS(linelist_clean, "data/clean/linelist_clean.rds")
saveRDS(contacts, "data/clean/contacts.rds")Sobre este documento
Contribuciones
- Anne Cori, Natsuko Imai, Finlay Campbell, Zhian N. Kamvar & Thibaut Jombart: Versión inicial
- José M. Velasco-España: Traducción de Inglés a Español
- Andree Valle-Campos: Ediciones menores
Contribuciones son bienvenidas vía pull requests.
